完全長cDNAライブラリーの受託合成 : 技術情報
□ 完全長cDNAライブラリーの技術概要
□Cap-Trapper™法
ダナフォームの完全長cDNAライブラリーは、理化学研究所で開発されたCap-Trapper™法により作成するものです。 少量のtotal RNAから高い完全長率(最大95%)の完全長cDNAライブラリーを作成します。
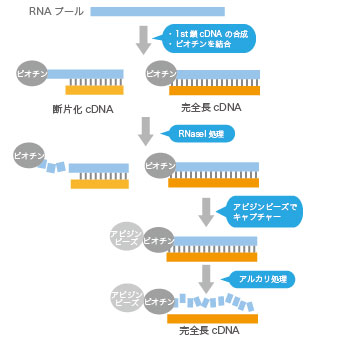
Cap-Trapper™法は、真核生物のmRNAに特有のCap構造をビオチン化した後、RNaseI処理により不完全長cDNAのCap構造を選択的に取り除きます。 残ったビオチン化したCap構造を有する完全長cDNAは、ストレプトアビジンをコートした磁気ビーズでキャプチャーします。 選択された完全長cDNAをアルカリ処理することによりビーズから溶出します。 最終的に、ビーズから分離され1本鎖となった第一鎖cDNAの第二鎖を合成することにより、完全長cDNAを選択的に得ることができます。
□ 論文
□技術に関する論文
-
P. Carninci et al, High-efficiency full-length cDNA cloning by biotinylated CAP trapper, Genomics, 37, 327?336 (1996)
-
P. Carninci et al. High efficiency selection of full-length cDNA by improved biotinylated cap trapper, DNA Res, 28, 61-66 (1997)
-
P. Carninci et al, Thermostabilization and thermoactivation of thermolabile enzymes by trehalose and its application for the synthesis of full length cDNA, PNAS 25, 520-524 (1998)
-
P. Carninci et al, High-efficiency full-length cDNA cloning, Methods Enzymol, 303, 19-44 (1999)
-
P. Carninci et al, E. Valen et al, Normalization and subtraction of cap-trapper-selected cDNAs to prepare full-length cDNA libraries for rapid discovery of new genes, Genome Res, 10, 1617-1630 (2000)
-
Y. Shibata et al, Cloning full-length, cap-trapper-selected cDNAs by using the single-strand linker ligation method, Biotechniques, 30, 1250-1254 (2001)
-
P. Carninci et al, Balanced-size and long-size cloning of full-length, cap-trapped cDNAs into vectors of the novel lambda-FLC family allows enhanced gene discovery rate and functional analysis, Genomics, 77, 79-90 (2001)
-
Y. Shibata et al, Removal of polyA tails from full-length cDNA libraries for high-efficiency sequencing, Biotechniques, 31, 1042-1044 (2001)
-
P. Carninci et al, Extra-long first-strand cDNA synthesis, Biotechniques, 32, 984-985 (2002)
-
Y. Shibata et al, Cytoplasmic RNA extraction from fresh and frozen mammalian tissue, Biotechniques, 33, 306-309 (2002)
-
P. Carninci et al, Targeting a complex transcriptome: the construction of the mouse full-length cDNA encyclopedia, Genome Res, 13, 1273-1289 (2003)
-
T. Hirozane-Kishikawa et al, Subtraction of cap-trapped full-length cDNA libraries to select rare transcripts, Biotechniques , 35, 510-518 (2003)