CAGE-seq受託解析 : 技術情報
□ 技術概要
CAGE(Cap Analysis Gene Expression)-seqは、理化学研究所(理研)とダナフォームが、遺伝子発現プロファイルやプロモーターを特定する新しいアプローチとして共同で開発した技術です。
CAGE-seqは、完全長cDNAの5'末端にアダプターをライゲーションさせる完全長cDNAライブラリー作製技術がベースになっており、これを次世代シーケンス解析に供します。 シーケンスリードをゲノム配列上でマッピングすることにより、転写開始点を同定することができます。 このように、CAGE-seqは、新規遺伝子の発見、ゲノムワイドな遺伝子発現解析、プロモーター解析の強力なツールとなります。
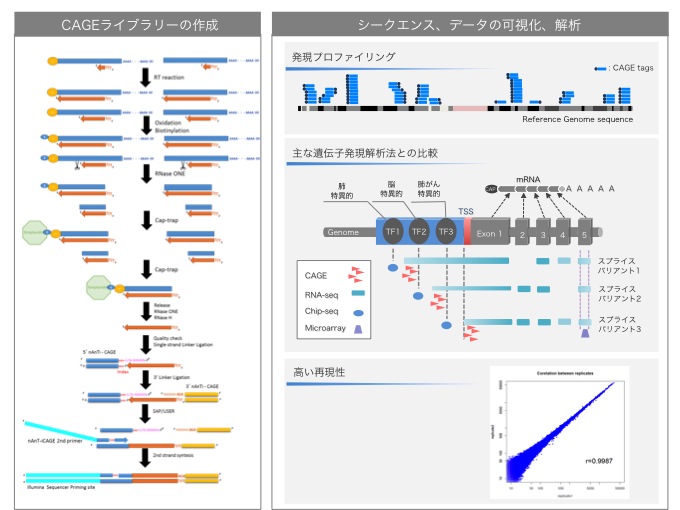
□他の発現解析技術との比較
遺伝子発現解析の手法は多くあり、それぞれに利点があります。 CAGE-seqと主な遺伝子発現解析の違いは、CAGE-seqがゲノムワイドに転写開始点をより正確に同定できるという点にあります。
CAGE-seqでは、プロモーター領域の詳細な解析ができるため、特定遺伝子の発現を、制御に関与したプロモーターごとに分けて解析することが可能となり、転写のシグナル伝達のカスケードを明らかにするなど、新しい視点に立ったゲノムアノテーションが可能になります。
| CAGE | RNA-seq | SAGE | マイクロアレイ | |
|---|---|---|---|---|
| 未知遺伝子を含む |
○ | ○ | ○ | × |
| 定量性/ダイナミックレンジ | ◎ ※PCR行程も無く、長さのバイアスも無い |
○ | ○ | △ |
| プロモーター部位の同定 | ◎ | △ | × | × |
| Transcription Factor Binding Motifの予測 | ◎ | △ | × ※5'末端はデータベース情報に依存 |
× ※5'末端はデータベース情報に依存 |
| Bidirectional enhancer RNAの同定 | ◎ | × | × | × |
| Alternative Exon1の同定 | ◎ | △ | × | × |
| 選択的スプライシングや 融合転写物など遺伝子の構造の同定 |
× | △ ※Sequence depthによる |
× | × |
| 全工程にかかる解析所要期間 | △ | △ | △ | ○ |
| サンプル調整難易度 | × ※8日間の行程 |
△ | △ | ◎ |
| データ解析ツール | △ | ○ | △ | ○ |
□ 論文
□FANTOM主要論文
□その他
- Young RS et al, The frequent evolutionary birth and death of functional promoters in mouse and human, Genome Res, 25, 1546-1557 (2015)
- Roy S et al, Redefining the transcriptional regulatory dynamics of classically and alternatively activated macrophages by deepCAGE transcriptomics, Nucleic Acids Res, 43(14), 6969-6982 (2015)
- Yu NY et al, Complementing tissue characterization by integrating transcriptome profiling from the Human Protein Atlas and from the FANTOM5 consortium, Nucleic Acids Res, 43(14), 6787-6798 (2015)
- Mina M et al, Promoter-level expression clustering identifies time development of transcriptional regulatory cascades initiated by ErbB receptors in breast cancer cells, Sci Rep, 5, 11999 (2015)
- Ramilowski JA et al, A draft network of ligand-receptor-mediated multicellular signalling in human, Nat Commun, 6, 7866 (2015)
- Aitken S et al, Transcriptional dynamics reveal critical roles for non-coding RNAs in the immediate-early response, PLoS Comput Biol, 11(4), e1004217 (2015)
- Haberle V et al, CAGEr: precise TSS data retrieval and high-resolution promoterome mining for integrative analyses, Nucleic Acids Res, 43(8), e51 (2015)
- Taguchi A et al, Characterization of Novel Transcripts of Human Papillomavirus Type 16 Using Cap Analysis Gene Expression Technology, Journal of Virology, 89(4), 2448-2452 (2015)
- Arner E et al, Gene regulation. Transcribed enhancers lead waves of coordinated transcription in transitioning mammalian cells, Science, 347(6225), 1010-1014 (2015)
- Joshi A et al, Transcription factor, promoter, and enhancer utilization in human myeloid cells, J Leukoc Biol, 97(5), 985-995 (2015)
- Fort A et al, Nuclear transcriptome profiling of induced pluripotent stem cells and embryonic stem cells identify noncoding loci resistant to reprogramming, Cell Cycle, 14(8), 1148-1155 (2015)
- Lizio M et al, Gateways to the FANTOM5 promoter level mammalian expression atlas, Genome Biol, 16, 22 (2015)
- Vitezic M et al, CAGE-defined promoter regions of the genes implicated in Rett Syndrome, BMC Genomics, 15, 1177 (2014)
- Verardo R et al, Specific mesothelial signature marks the heterogeneity of mesenchymal stem cells from high-grade serous ovarian cancer, Stem Cells, 32(11), 2998-3011 (2014)
- Grison A et al, Mesencephalic dopaminergic neurons express a repertoire of olfactory receptors and respond to odorant-like molecules, BMC Genomics, 15, 729 (2014)
- Fort A et al, Deep transcriptome profiling of mammalian stem cells supports a regulatory role for retrotransposons in pluripotency maintenance, Nature Genetics, 46(6), 558-566 (2014)
- Hasegawa A et al, MOIRAI: a compact workflow system for CAGE analysis, BMC Bioinformatics, 15, 144 (2014)
- Yamaga R et al, Systemic identification of estrogen-regulated genes in breast cancer cells through cap analysis of gene expression mapping, Biochem Biophys Res Commun, 447(3), 531-536 (2014)
- Prasad P et al, High-throughput transcription profiling identifies putative epigenetic regulators of hematopoiesis, Blood, 123(17), e46-e57 (2014)
- Ohmiya H et al, RECLU: a pipeline to discover reproducible transcriptional start sites and their alternative regulation using capped analysis of gene expression (CAGE), BMC Genomics, 15, 269 (2014)
- Kawaji H et al, Comparison of CAGE and RNA-seq transcriptome profiling using a clonally amplified and single molecule next generation sequencing, Genome Res, 24(4), 708-717 (2014)
- Rönnerblad M et al, Analysis of the DNA methylome and transcriptome in granulopoiesis reveals timed changes and dynamic enhancer methylation, Blood, 123(17), e79-e89 (2014)
- Motakis E et al, Redefinition of the human mast cell transcriptome by deep-CAGE sequencing, Blood, 123(17), e58-e67 (2014)
- Schmidl C et al, The enhancer and promoter landscape of human regulatory and conventional T cell subpopulations, Blood, 123(17), e68-e78 (2014)
- Schmidl C et al, Transcription and enhancer profiling in human monocyte subsets, Blood, 123(17), e90-e99 (2014)
- Morikawa H et al, Differential roles of epigenetic changes and Foxp3 expression in regulatory T cell-specific transcriptional regulation, Proc Natl Acad Sci USA, 111(14), 5289-5294 (2014)
- Severin J et al, Interactive visualization and analysis of large-scale NGS data-sets using ZENBU, Nature Biotechnology, 32(3), 217-219 (2014)
- Rye M et al, Chromatin states reveal functional associations for globally defined transcription start sites in four human cell lines, BMC Genomics, 15, 120 (2014)
- Murata M et al, Detecting expressed genes using CAGE, Methods Mol Biol, 1164, 67-85 (2014)
- Pascarella G et al, NanoCAGE analysis of the mouse olfactory epithelium identifies the expression of vomeronasal receptors and of proximal LINE elements, Front Cell Neurosci, 8, 41 (2014)
- Nepal C et al, Dynamic regulation of the transcription initiation landscape at single nucleotide resolution during vertebrate embryogenesis, Genome Res, 23(11), 1938-1950 (2013)
- Sompallae R et al, Sompallae RA comprehensive promoter landscape identifies a novel promoter for CD133 in restricted tissues, cancers, and stem cells, Front Genet, 4, 209 (2013)
- Sompallae R, A comprehensive promoter landscape identifies a novel promoter for CD133 in restricted tissues, cancers, and stem cells, Front Genet, 4, 209 (2013)
- Harbers M et al, Comparison of RNA- or LNA-hybrid oligonucleotides in template-switching reactions for high-speed sequencing library preparation, BMC Genomics, 14, 665 (2013)
- Tang DT et al, Suppression of artifacts and barcode bias in high-throughput transcriptome analyses utilizing template switching, Nucleic Acids Res, 41(3), e44 (2013)
- ENCODE Project Consortium, An integrated encyclopedia of DNA elements in the human genome, Nature, 489(7414), 57-74 (2012)
- Derrien T et al, The GENCODE v7 catalog of human long noncoding RNAs: analysis of their gene structure, evolution, and expression, Genome Res, 22(9), 1775-1789 (2012)
- Djebali S et al, Landscape of transcription in human cells, Nature, 489(7414), 101-108 (2012)
- Itoh M et al, Automated workflow for preparation of cDNA for cap analysis of gene expression on a single molecule sequencer, PLoS One, 7(1), e30809 (2012)
- Takahashi H et al, CAGE (cap analysis of gene expression): a protocol for the detection of promoter and transcriptional networks, Methods Mol Biol, 786, 181-200 (2014)
- Schroder K et al, Conservation and divergence in Toll-like receptor 4-regulated gene expression in primary human versus mouse macrophages, Proc Natl Acad Sci USA, 109(16), E944-E953 (2012)
- Plessy C et al, Promoter architecture of mouse olfactory receptor genes, Genome Res, 22(3), 486-497 (2012)
- Takahashi H et al, 5' end-centered expression profiling using cap-analysis gene expression and next-generation sequencing, Nat Protoc, 7, 542-561 (2012)
- Chien CH et al, Identifying transcriptional start sites of human microRNAs based on high-throughput sequencing data, Nucleic Acids Res, 39(21), 9345-9356 (2011)
- Kanamori-Katayama M et al, Unamplified cap analysis of gene expression on a single-molecule sequencer, Genome Res, 21(7), 1150-1159 (2011)
- Hoskins RA et al, Genome-wide analysis of promoter architecture in Drosophila melanogaster, Genome Res, 21(2), 182-192 (2011)
- Salimullah M et al, NanoCAGE: a high-resolution technique to discover and interrogate cell transcriptomes, Cold Spring Harb Protoc, 2011(1), pdb.prot5559 (2011)
- Schaefer U et al, High sensitivity TSS prediction: estimates of locations where TSS cannot occur, PLoS One, 5(11), e13934 (2010)
- Vitezic M et al, Building promoter aware transcriptional regulatory networks using siRNA perturbation and deepCAGE, Nucleic Acids Res, 38(22), 8141-8148 (2010)
- Hestand MS et al, Tissue-specific transcript annotation and expression profiling with complementary next-generation sequencing technologies, Nucleic Acids Res, 38(16), e165 (2010)
- Plessy C et al, Linking promoters to functional transcripts in small samples with nanoCAGE and CAGEscan, Nat Methods, 7(7), 528-534 (2010)
- Atanur SS et al, The genome sequence of the spontaneously hypertensive rat: Analysis and functional significance, Genome Res, 20(6), 791-803 (2010)
- FANTOM Consortium et al, The transcriptional network that controls growth arrest and differentiation in a human myeloid leukemia cell line, Nat Genet, 41(5), 553-62 (2009)
- Faulkner GJ et al, The regulated retrotransposon transcriptome of mammalian cells, Nat Genet, 41(5), 563-571 (2009)
- Balwierz PJ et al, Methods for analyzing deep sequencing expression data: constructing the human and mouse promoterome with deepCAGE data, Genome Biol, 10(7), R79 (2009)
- Valen E et al, Genome-wide detection and analysis of hippocampus core promoters using DeepCAGE, Genome Res, 19(2), 255-265 (2009)
- Hofmann O et al, Genome-wide analysis of cancer/testis gene expression, Proc Natl Acad Sci USA, 105(51), 20422-20427 (2008)
- ENCODE Project Consortium et al, Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project, Nature, 447(7146), 799-816 (2007)
- Shimokawa K et al, Large-scale clustering of CAGE tag expression data, BMC Bioinformatics, 8, 161 (2007)
- Kawaji H et al, Dynamic usage of transcription start sites within core promoters, Genome Biol, 7(12), R118 (2006)
- Carninci P et al, Genome-wide analysis of mammalian promoter architecture and evolution, Nat Genet, 38(6), 626-635 (2006)
- Kodzius R et al, CAGE: cap analysis of gene expression, Nat Methods, 3(3), 211-222 (2006)
- Shiraki T et al, Cap analysis gene expression for high-throughput analysis of transcriptional starting point and identification of promoter usage, Proc Natl Acad Sci USA, 100(26), 15776-15781 (2003)
- Carninci P et al, High-efficiency full-length cDNA cloning by biotinylated CAP trapper, Genomics, 37(3), 327-336 (1996)