株式会社ダナフォーム
 |
|
|
|
| mRNA/ncRNAなどの発現を転写開始点ごとに定量解析 | |
|
|
|
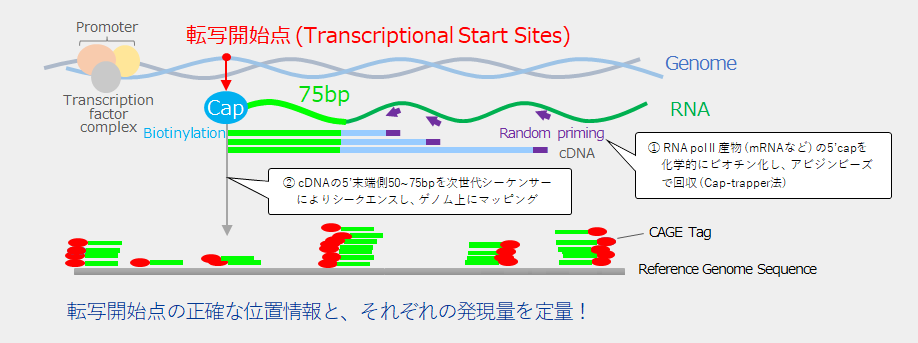
CAGE : 転写開始点をとらえるトランスクリプトーム解析
CAGE(Cap Analysis of Gene Expression) は、RNAポリメラーゼII 産物のCap構造を捕捉するCap-trapper法によりRNAの5’末端の塩基配列を決定、ゲノム上にマッピングする技術です。転写開始点の位置を1塩基単位、かつゲノムワイドに同定することで、各転写産物のプロモーター領域や発現量を的確にとらえた発現プロファイルを可能にします。
CAGEは転写のシグナル伝達のカスケードを明らかにするなど、新しい視点に立ったゲノムアノテーション研究の強力なツールとなります。
CAGEは転写のシグナル伝達のカスケードを明らかにするなど、新しい視点に立ったゲノムアノテーション研究の強力なツールとなります。
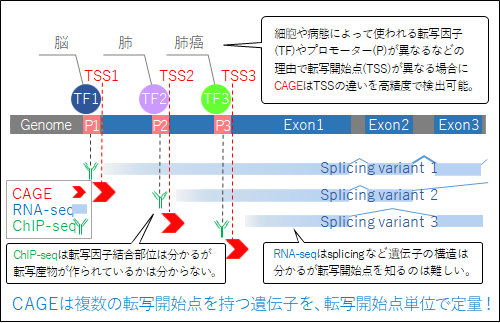
CAGEと主な遺伝子発現解析の違い
|
 |
||||||||||||||
|
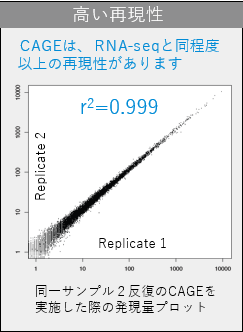 |
| 1. PCRの工程が無く、長さのバイアスも受けない 2. 5’末端はデータベース情報に依存 3. Sequence depthによる |
4. 8日間の工程 5. 1-2ヵ月 |
解析例 1
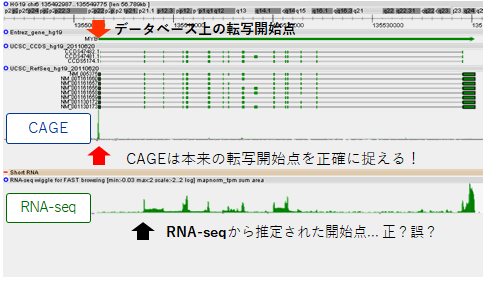
遺伝子の複数の転写開始点を1bpの解像度で解析
例1. 本来の転写開始点がクリアに分かる! |
例2. 新規バイオマーカー探索に有用! |
 |
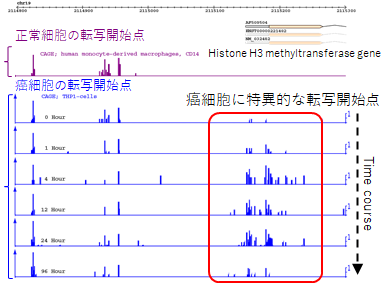 |
解析例 2
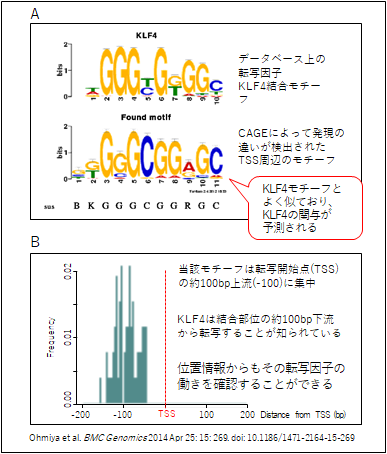
転写開始点周辺の転写因子結合モチーフの探索
例1. 細胞間で発現が異なる転写開始点の近傍モチーフ探索 |
例2.TSSからモチーフまでの位置をとらえる! |
||||||||||||||||||||||||||||||||||||||||||||||||
間葉系幹細胞(Control)と脂肪前駆細胞(Case)でCAGE発現に違いが見られ
た転写開始点の近傍で見つかった転写因子結合モチーフ候補
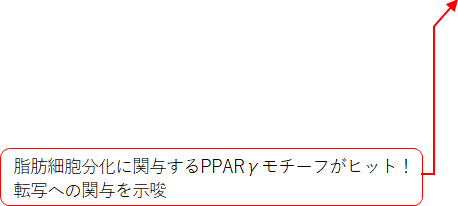
|
 |
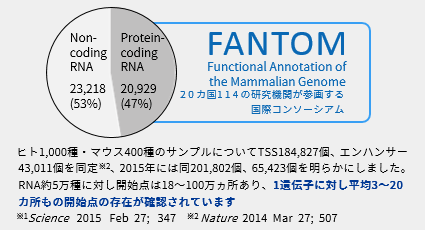
理化学研究所FANTOMプロジェクト
| 20カ国、100以上の研究機関が参加する国際研究コンソーシアムです。ヒトの主要な組織、初代培養細胞、細胞株をCAGEを用いて解析し、201,802個のプロモーターと65,423個のエンハンサーを同定することに成功しました※1。その役割はトランスクリプトーム解析の分野を軸に発展・拡大しており、得られた知見を基礎・応用の両面で有用なリソースとして公開しています。 | |||||||||||||||||||||||||||||||||
|
 |
||||||||||||||||||||||||||||||||
CAGEを用いた主な論文
- CXCL4/PF4 is a predictive biomarker of cardiac differentiation potential of human induced pluripotent stem cells.
Ohashi F, et al. Sci. Rep. 2019 Mar 15;9:4638. doi: 10.1038/s41598-019-40915-w. - Promoter-level transcriptome in primary lesions of endometrial cancer identified biomarkers associated with lymph node metastasis.
Yoshida E, et al. Sci. Rep. 2017 Oct 26;7:14160. doi: 10.1038/s41598-017-14418-5. - An atlas of human long non-coding RNAs with accurate 5’ ends.
Hon CC, et al. Nature. 2017 Mar 9;543(7644):199-204. doi: 10.1038/nature21374. - Single-Nucleotide Resolution Mapping of Hepatitis B Virus Promoters in Infected Human Livers and Hepatocellular Carcinoma.
Altinel K, et al. J Virol. 2016 Nov 14;90(23):10811-10822. - Reduced expression of APC-1B but not APC-1A by the deletion of promoter 1B is responsible for familial adenomatous polyposis.
Yamaguchi K, et al. Sci. Rep. 2016 May 24;6:26011. doi: 10.1038/srep26011. - Enhanced Identification of Transcriptional Enhancers Provides Mechanistic Insights into Diseases.
Murakawa Y, et al. Trends Genet. 2016 Feb;32(2):76-88. doi: 10.1016/j.tig.2015.11.004. - DeepCAGE Transcriptomics Reveal an Important Role of the Transcription Factor MAFB in the Lymphatic Endothelium.
Dieterich LC, et al. Cell Rep. 2015 Nov 17;13(7):1493-504 - Nuclear transcriptome profiling of induced pluripotent stem cells and embryonic stem cells identify non-coding loci resistant to reprogramming.
Fort A, et al. Cell Cycle. 2015;14(8):1148-55. doi: 10.4161/15384101.2014.988031. - Transcribed enhancers lead waves of coordinated transcription in transitioning mammalian cells.
Arner E, et al. Science 2015 Feb 27;347(6225):1010-4. doi126/science.1259418. - Deep transcriptome profiling of mammalian stem cells supports a regulatory role for retrotransposons in pluripotency maintenance
Fort A, et al. Nat. Genet. 2014 Jun 28;46:558-566. doi:10.1038/ng.2965. - Two independent transcription initiation codes overlap on vertebrate core promoters.
Haberle V, et al. Nature 2014 Mar 20; 507(7492):381-385. doi: 10.1038/nature12974.
基本サービス
|
 |
|
 |
|
|||||||||||||||||||||||||||||||||||||||||||||
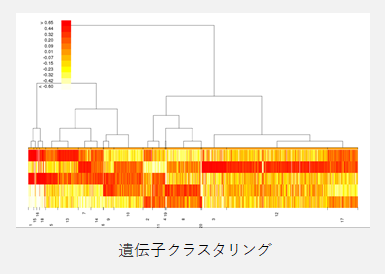 |
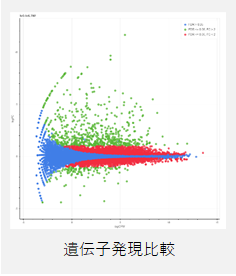 |
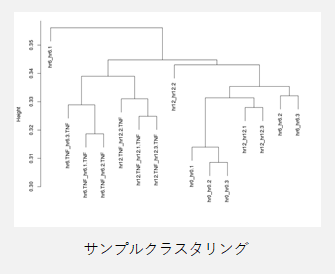 |
価 格
|
|
|||||||||||||||||||||||||
|
株式会社ダナフォーム
〒230-0051 神奈川県横浜市鶴見区鶴見中央 2-6-29
アスク・サンシンビル 3F TEL : 045-508-1539 FAX : 045-510-0608 E-mail : contact@dnaform.jp URL : http://dnaform.jp/ja/ |
|
| CAGE-V02-07-00-210714 |
|